近日,中国农业科学院棉花研究所生物信息与分子设计中心联合华中农业大学信息学院和新疆农垦科学院棉花所等多家单位通过搜集和整合棉花基因组、转录组、变异组、表观遗传、表型组和代谢组等6个组学数据,构建出了棉花多组学数据库(CottonMD),为棉花遗传育种研究提供了重要的数据资源和分析平台。相关研究成果以“CottonMD: a multi-omics database for cotton biological study”为题发表在国际权威学术期刊《核酸研究(Nucleic Acids Research)》(IF=19.16,Q1)上。
棉花是世界上重要的天然纤维作物,我国是世界上最重要的棉花生产国和原棉消费国,棉花事关国计民生。当前我国主要通过传统遗传育种选育棉花新品种,存在挖掘候选基因的精度和效率都较低的问题。近年来,逐渐发展成熟的多组学技术提供了更多维度的信息,为解决传统遗传育种方法中存在的问题、加速实现精准育种提供了新的途径。为发挥多组学技术在育种中的价值,水稻、玉米、高粱等多个主要作物的多组学数据库平台已相继被建立,但目前仍缺少一个更为综合型的棉花多组学数据库平台。
研究人员通过整合25个棉花基因组、76个组织样本的转录组、5个物种的表观遗传学、4180个样本的群体遗传变异数据、20个表型和768个代谢物含量等公共多组学数据,构建了目前国际上最为系统和全面的棉花多组学数据库。该数据库包含大量来自不同组学的信息,用户可以通过输入基因ID或物理位置信息来检索基因相关的多组学信息。这些功能为用户快速准确地理解基因的功能提供了快速方便的工具。在该数据库中,研究者利用全基因组关联分析(GWAS)、表达数量性状位点定位(eQTL)、孟德尔随机化(SMR)和共定位分析等多组学关联分析方法对不同组学的棉花数据进行关联,并将分析结果和工具整合到数据库中以方便用户查询、分析和利用。此外,该数据库提供了丰富的棉花多组学数据和多种在线多组学分析以及种质资源管理工具。该数据库所有模块均支持25个已发表棉花基因组的基因ID进行搜索、浏览和数据下载,以服务全球范围内的相关研究工作。
据悉,中棉所生物信息中心于2022年初通过整合多个棉属物种基因组、转录组、变异位点和基因互作网络等信息,构建了界面友好、交互性强的棉花多组学数据库GRAND。目前发布的CottonMD是一个更为综合型的棉花多组学数据库平台,也是首个利用多种关联分析方法挖掘“变异-基因表达-表型”之间关联信息并提供相应数据可视化查询的数据库,为棉花分子设计育种研究提供了重要的数据资源和分析平台。
中棉所杨作仁研究员和华中农业大学杨庆勇教授为论文共同通讯作者,中棉所杨召恩研究员等参与研究。该研究得到国家重点研发计划(2021YFF1000100)、河南省自然科学基金(212300410093)、海南省崖州湾种子实验室生物信息学开发平台(JBGS-B21HJ0001)等项目的资助。
数据库链接:http://grand.cricaas.com.cn/home
原文链接:https://doi.org/10.1093/nar/gkac863
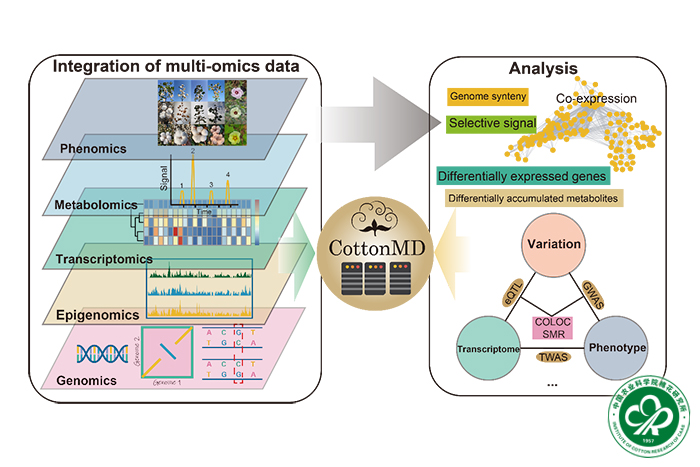
CttonMD数据库的构建和功能